다음과 같은 유전자 리스트를 가지고 있을 때,
CPT2
CPT1A
FACL1
EHHADH
사람에서 이와 관련 있는 pathway에는 무엇이 있는지 KEGG Mapper를 사용하여 찾는 방법을 소개하고자 한다.
KEGG (Kyoto Encyclopedia of Genes and Genomes) Mapper를 이용하면 input gene list가 pathway의 어느 곳에 나타나는지 색깔로 표시할 수 있다 (ex. upregulation gene을 빨간색으로 표시하는 등).
하지만 input으로 위와 같은 Gene symbol (CTP2, CPT1A, ... )가 아니라 KEGG ID를 받기 때문에 Symbol을 KEGG ID로 먼저 변환해줘야 한다. 이는 다음 사이트에서 가능하다.
https://biodbnet-abcc.ncifcrf.gov/db/db2db.php
bioDBnet - Biological Database Network
db2db allows for conversions of identifiers from one database to other database identifiers or annotations. To use db2db select the input type of your data, changing the input type automatically changes the output options to the ones specific for the input
biodbnet-abcc.ncifcrf.gov
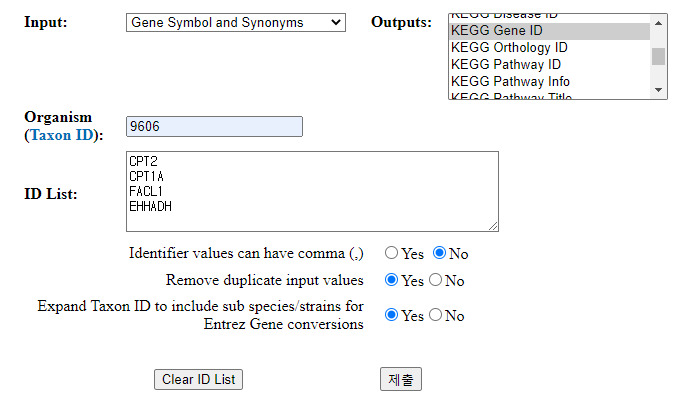
위와 같이 입력하면 Gene Symbol (대표 symbol 외에 synonyms까지 포함)을 KEGG Gene ID로 변환할 수 있다. 이때 Taxon ID는 9606을 적었는데, 이는 사람(Homo sapiens)을 나타낸다.
제출하면 다음과 같은 결과를 얻을 수 있다.

이를 KEGG Mapper에 입력한다.
https://www.genome.jp/kegg/mapper/color.html
KEGG Mapper Color
Color tool The Color tool searches various KEGG objects, including genes, KOs, EC numbers, metabolites and drugs, against KEGG pathway maps. Found objects may be marked in any combination of background and foreground colors. Usage Query data format KEGG id
www.genome.jp

Search mode는 hsa를 해줘야 한다.
Exec을 누르면 다음과 같은 결과를 얻을 수 있다.

위의 pathway들이 우리가 입력한 4개의 유전자와 관련있는 pathway들이다. Pathway 별로 4개의 유전자 중 몇 개를 포함하고 있는지 숫자로 알려준다.
'Fatty acid degradation'을 클릭해보았다.
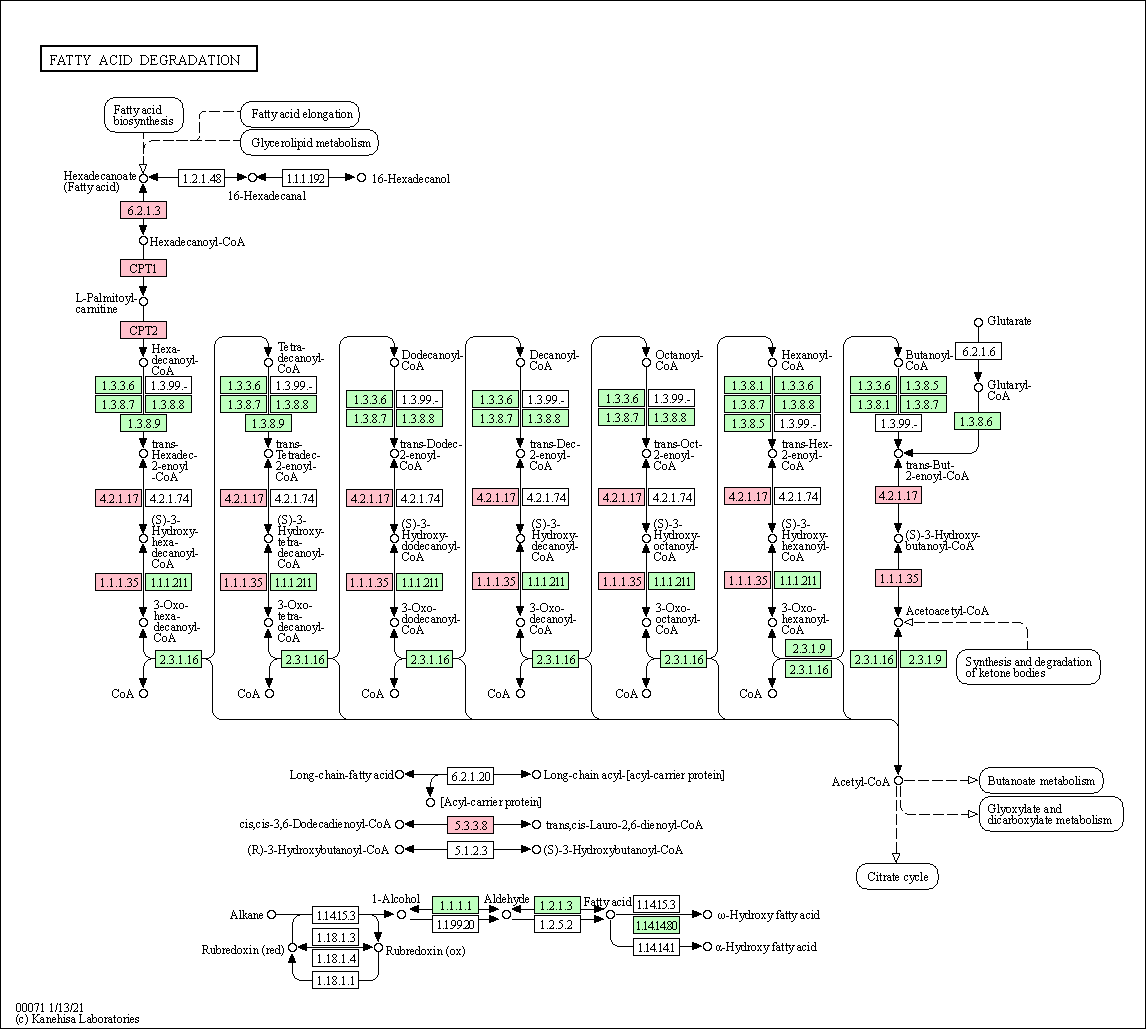
입력한 유전자만 분홍색으로 강조되어 있는 모습을 확인할 수 있다.
'Bioinformatics > Transcriptomics' 카테고리의 다른 글
| KEGG 데이터베이스에서 유전자 검색 | KEGG 데이터베이스 구조 (0) | 2021.08.17 |
|---|---|
| [Python] Find Open Reading Frame (ORF) | 파이썬 코드 (0) | 2021.06.14 |
| Gene Expression Quantification | RPM, RPKM, FPKM, TPM 공식 | 파이썬 코드 (0) | 2021.06.14 |
| [Prodigal] 원핵생물의 유전자 예측 프로그램 (Prokaryotic Gene prediction) (16) | 2021.05.07 |